Benchmark des filesystems sur Dedibox v1, v2, v3, sur Ubuntu et Debian

J'ai commandé récemment trois dedibox v3 : deux pour l'AFPY, une pour Gorfou, afin de remplacer les anciennes. Cette dedibox v3 est sortie il y a peu, et semble à première vue être intéressante : pour deux fois moins cher, on a plus de mémoire vive, plus d'espace disque, un CPU plus rapide, avec 64bits et virtualisation, et plus de bande passante. J'ai réalisé que j'avais accès aux trois générations de dedibox : les deux serveurs principaux AFPY et Gorfou sont des dedibox v1, et le serveur secondaire de l'AFPY, qui servait pour les buildbots, est une dedibox v2 (mise à disposition par Toonux).
J'en ai profité pour lancer un test de filesystem en utilisant iozone.
Voici les machines utilisées :
| nom | génération | système | archi | filesystem |
|---|---|---|---|---|
| py | dedibox v1 | Debian | Lenny 5.0 | 32 bits |
| cody | dedibox v1 | Ubuntu 10.04 Lucid | 32 bits | ext3 |
| boa | dedibox v2 | Debian 5.0 Lenny | 32 bits | ext3 |
| nouvelle | cody | dedibox v3 Ubuntu 10.04 Lucid | 64 bits | ext3 |
| nouvelle | boa | dedibox v3 Ubuntu 10.04 Lucid | 64 bits | ext3 |
| nouvelle | py | dedibox v3 Debian 6.0 Squeeze | 64 bits | ext3 |
J'ai juste lancé iozone -a (mode automatique) deux fois sur chaque serveur (1 seule fois sur cody).
Le résultat de iozone est un grand tableau de nombres illisible. Plutôt que d'ouvrir le résultat dans OpenOffice, j'ai trouvé que c'était une bonne occasion de jouer avec NumPy et Matplotlib pour extraire les données et tracer des résultats comparatifs en 3D.
Je tiens à signaler que les mesures n'ont pas été faites dans des conditions idéales, et que je n'ai pas cherché à modifier les options d'iozone. Si vous voulez comprendre les valeurs, reportez-vous à la doc d'iozone.
Voici les résultats :
Comparatif Dedibox V1 / V2 / V3
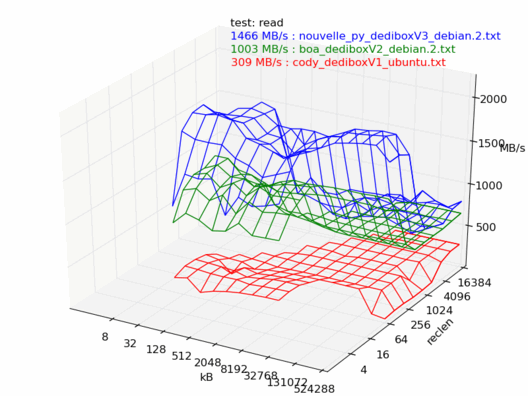
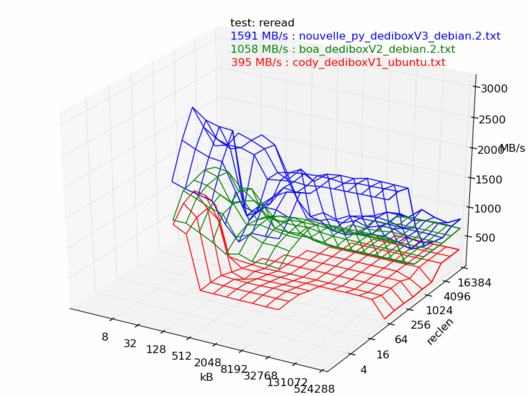
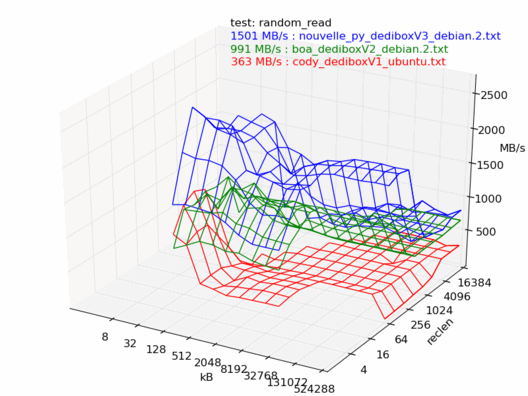
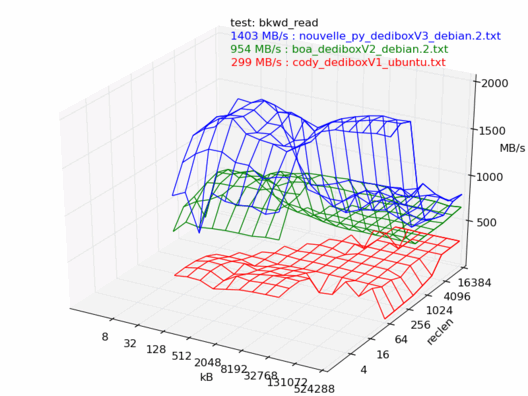
Lecture
En lecture, la dedibox v3 est la plus rapide des trois, il n'y a aucun doute.
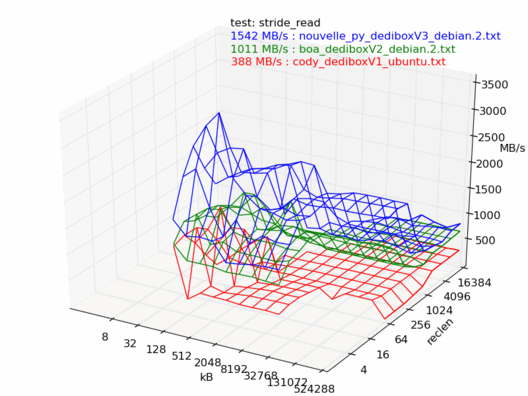
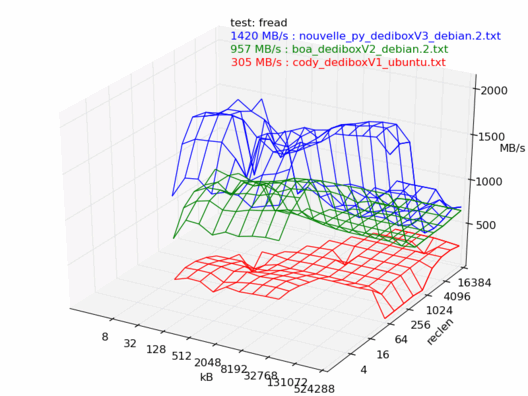
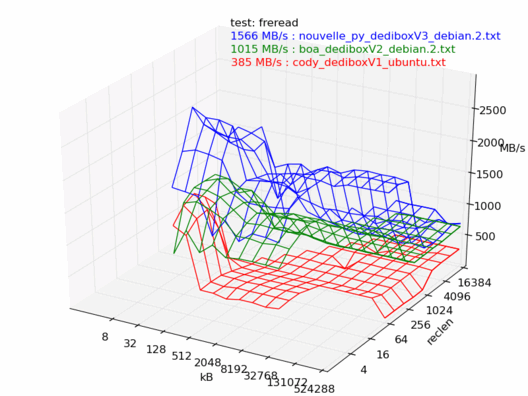
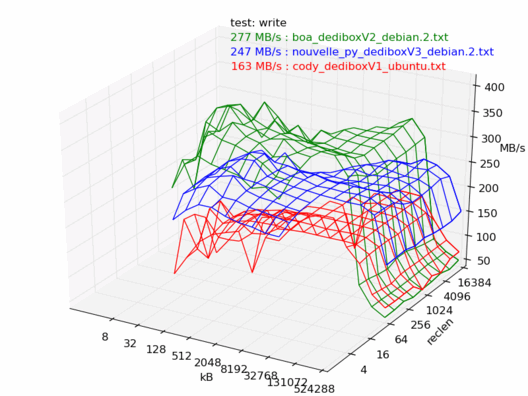
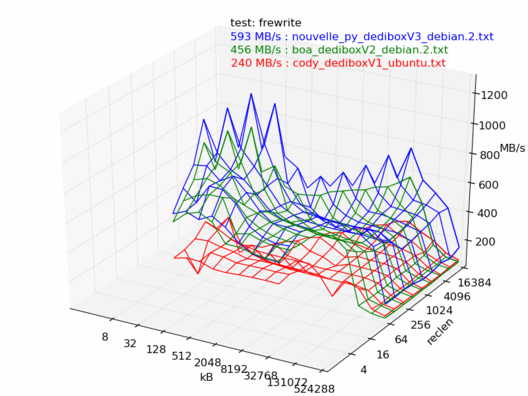
Écriture
En écriture, c'est un peu moins clair : la dedibox v3 est plus rapide dans 4 tests sur 6.
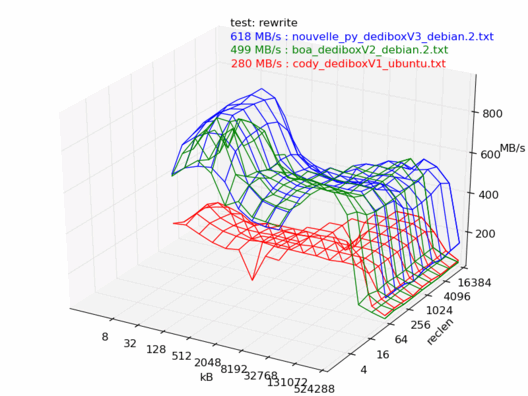
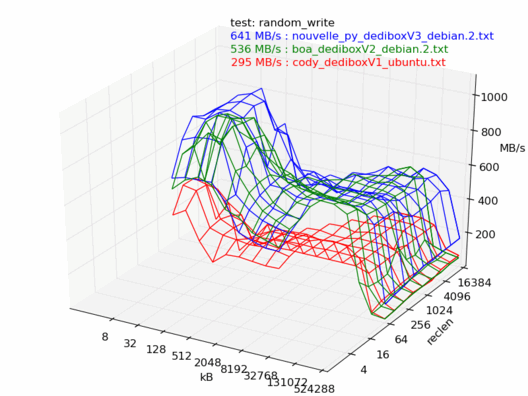
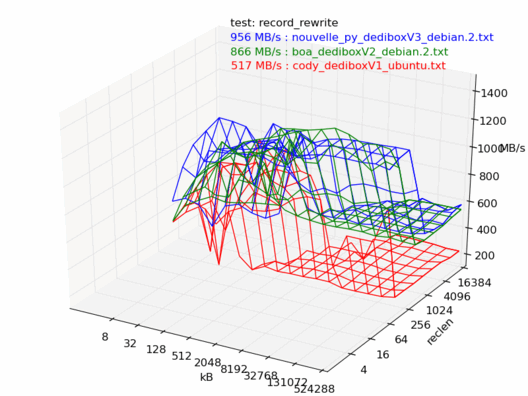
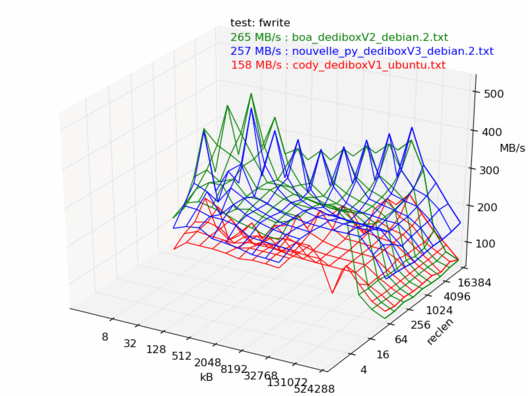
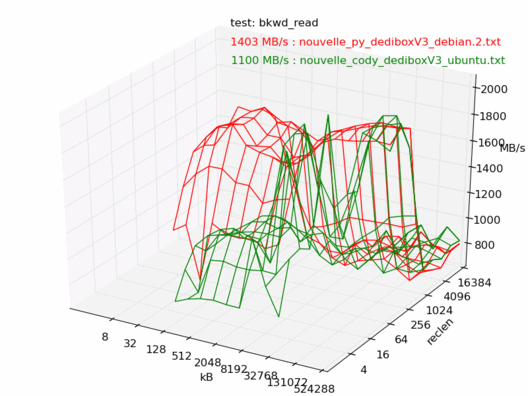
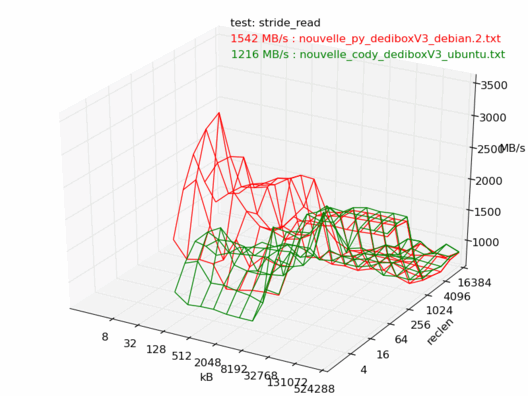
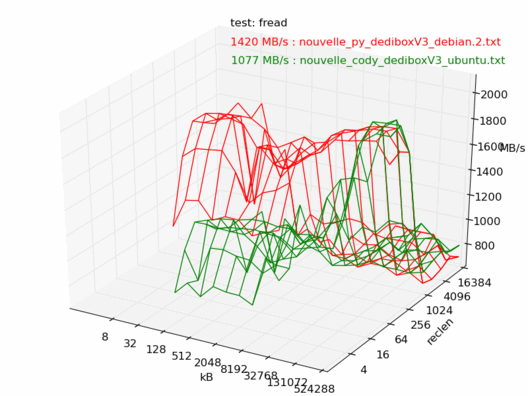
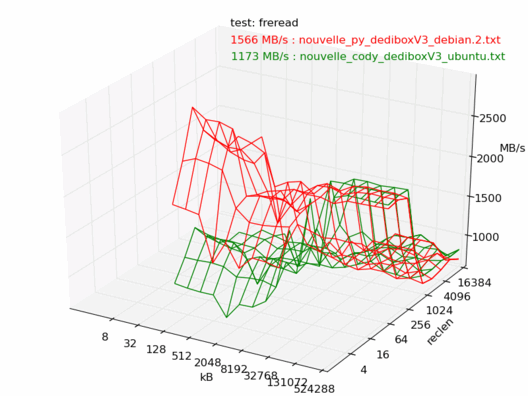
Comparatif Ubuntu / Debian
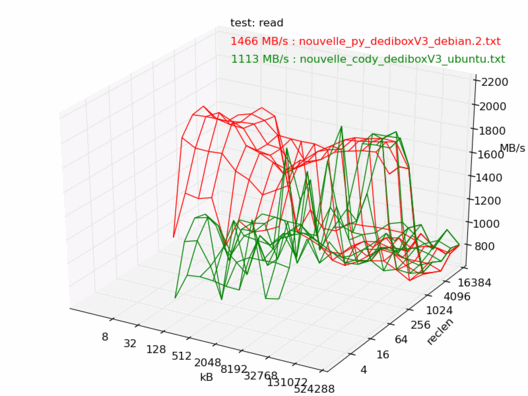
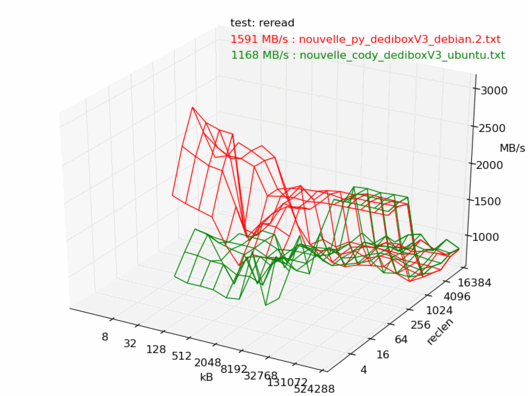
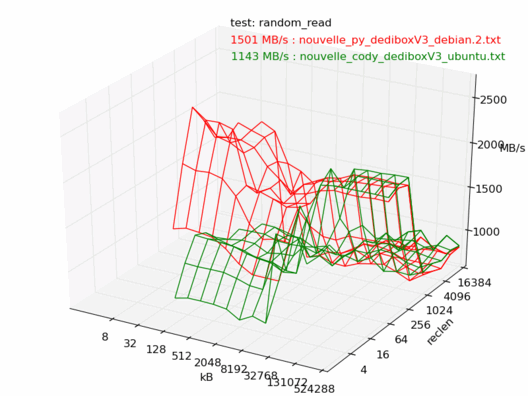
Voyons maintenant la différence entre Debian et Ubuntu. Ici les machines sont censées être les mêmes, sont toutes les deux vides et non utilisées, et sont installées avec les dernières versions de Debian 6.0 et Ubuntu 10.04. Les noyaux sont tous les deux des 2.6.32.
On note une très forte différence entre les deux : la machine sous Debian est beaucoup plus rapide.
Lecture
Écriture
Bizarre, mais même constat, la dedibox sous Debian est en moyenne une fois et demi plus rapide que celle avec Ubuntu.
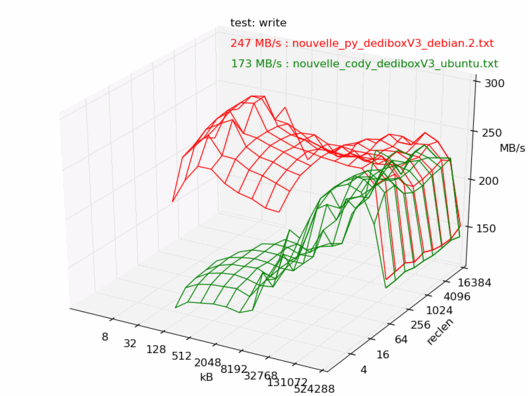
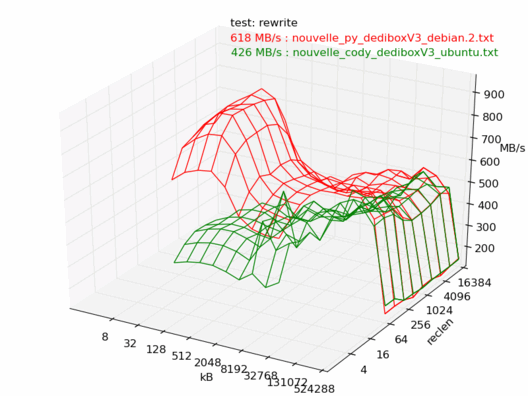
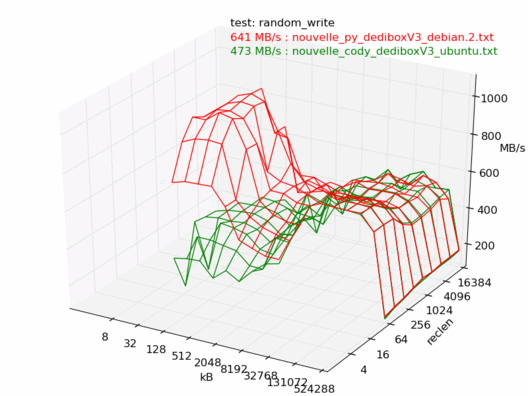
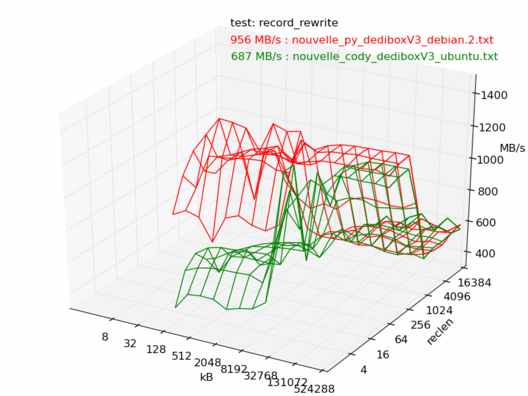
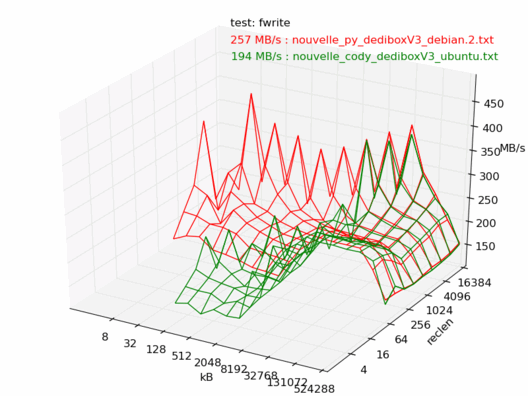
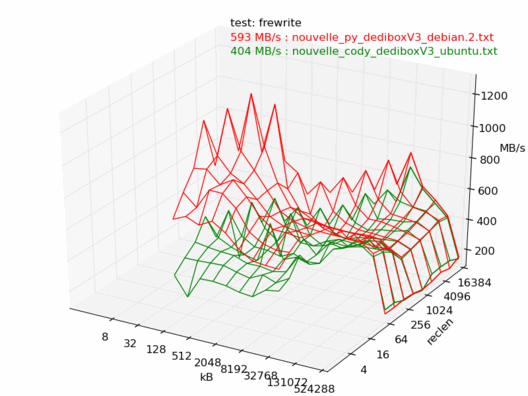
Je me garderai de faire des commentaires, vu les conditions dans lesquelles ces tests ont été réalisés, mais ça mériterait de creuser un peu...
Script d'extraction
Si vous voulez reproduire les graphiques, voici comment faire.
Il faut récupérer la sortie de iozone -a en ne gardant que le tableau, puis changer l'en-tête du tableau pour qu'il tienne sur une seule ligne, et sauver ça dans un fichier texte. Ensuite il faut installer Matplotlib 1.0. La version 0.99 fournie avec Ubuntu 10.04 ne convient pas, il faut compiler la version 1.0. Je vous laisse le doux plaisir de découvrir les dépendances à installer.
$ virtualenv sandbox
$ sandbox/bin/pip install install http://sourceforge.net/projects/matplotlib/files/matplotlib/matplotlib-1.0/matplotlib-1.0.0.tar.gz/downloadEnsuite voici le script qui a permis d'extraire et tracer les données, que j'appelle bench.py :
#!/usr/bin/env python
# coding: utf-8
import sys
import pylab, numpy as np
from numpy import log2
from mpl_toolkits.mplot3d import axes3d
tests = {}
files = sys.argv[1:]
pylab_colors = ('r', 'g', 'b', 'y', 'm', 'y', 'k')
assert len(files) <= len(pylab_colors)
colors = dict([(f, pylab_colors[i]) for i, f in enumerate(files)])
for k, filename in enumerate(files):
with open(filename) as f:
tests[filename] = {}
tests[filename]['titles'] = f.readline().split()[2:]
tests[filename]['raw'] = np.loadtxt(filename, dtype=int, skiprows=1)
for i, testname in enumerate(tests[filename]['titles']):
tests[filename][testname] = {}
data = tests[filename][testname]['data'] = np.zeros((20, 15))
data[:] = np.nan
raw = tests[filename]['raw']
data[log2(raw[:,0]).astype(int), log2(raw[:,1]).astype(int)] = raw[:, i+2]
data /= 1024.0
tests[filename][testname]['mean'] = data[-np.isnan(data)].mean()
tests[filename][testname]['max'] = data[-np.isnan(data)].max()
axes = {}
X, Y = np.mgrid[0:20,0:15]
for testname in tests.values()[0]['titles']:
ax = axes3d.Axes3D(pylab.figure())
ax.w_xaxis.set_ticks(range(3,21,2))
ax.w_yaxis.set_ticks(range(2,15,2))
ax.w_xaxis.set_ticklabels(2**np.arange(3,21,2))
ax.w_yaxis.set_ticklabels(2**np.arange(2,15,2))
ax.set_xlabel('kB')
ax.set_ylabel('reclen')
ax.set_zlabel('MB/s')
means = dict([(filename, tests[filename][testname]['mean']) for filename in files])
maxs = max([tests[filename][testname]['max'] for filename in files])
ax.text(X.min(), 15, maxs, 'test: ' + testname, color='k')
for i, filename in enumerate(sorted(means, key=lambda x:-means[x])):
i+=1
Z = tests[filename][testname]['data']
c = colors[filename]
ax.plot_wireframe(X, Y, Z, color=c)
ax.text(X.min(), 15, maxs*(1-0.07*i),
str(int(means[filename])) + " MB/s : " + filename, color=c)
#pylab.gcf().set_size_inches(6,4)
pylab.savefig(testname + '.png')
pylab.show()Pour le lancer avec la nouvelle version de matplotlib, il suffit d'utiliser le Python de la sandbox. Le script prend en paramètres les fichiers contenant les résultats iozone
$ ./sandbox/bin/python bench.py resultat1.txt resultat2.txt resultat3.txtIl est possible d'écrire un script beaucoup plus propre, mais je voulais juste m'obliger à le faire avec Numpy et Matplotlib et sans y passer trop de temps non plus. Notez qu'une des lignes du script utilise une affectation grâce au Fancy Indexing de Numpy:
data[log2(raw[:,0]).astype(int), log2(raw[:,1]).astype(int)] = raw[:, i+2]